Kunstig intelligens (AI) er med til at gentegne stamtræet af virus. Forudsagte proteinstrukturer vha AlphaFold og chatbot-inspirerede "Protein sprogmodeller" har afsløret overraskende forbindelser i en familie af vira, der inkluderer patogener, der inficerer mennesker og nye trusler.
En stor del af forskernes forståelse vedr viral udvikling er baseret på sammenligning af genomer. Den lynhurtige udvikling af vira, især dem med RNA-genomer, og deres tilbøjelighed til at erhverve genetisk materiale fra andre organismer viser dog, at genetiske sekvenser kan skjule dybere og fjernere forhold mellem vira, som kan variere afhængigt af det gen, der undersøges.
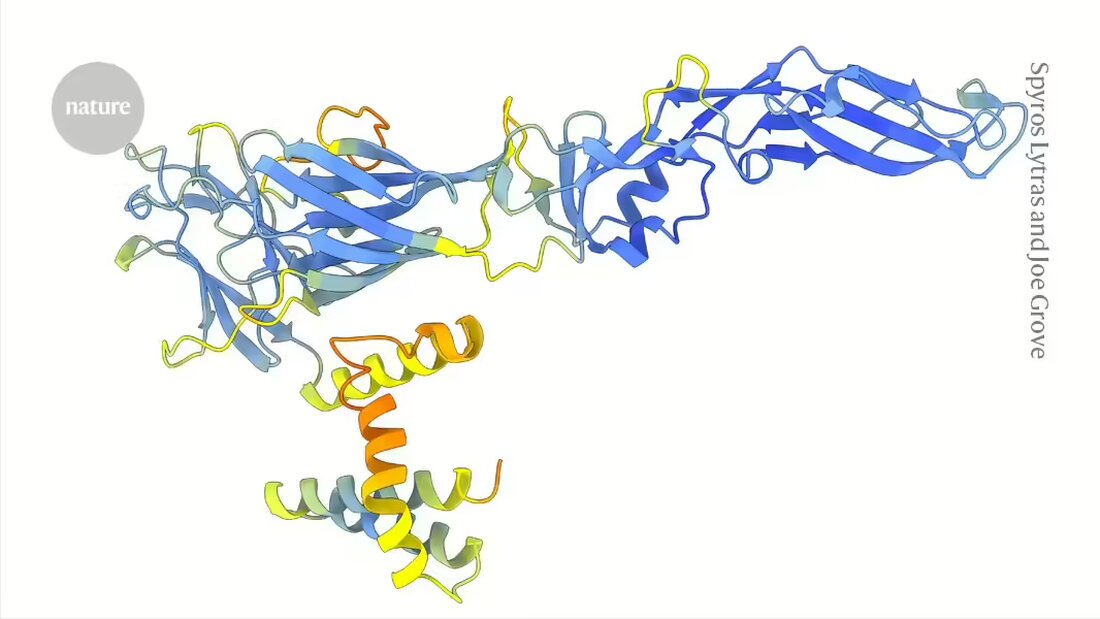
I modsætning hertil har formerne eller strukturerne af proteinerne kodet af virale gener en tendens til at ændre sig langsomt, hvilket gør det muligt at opdage disse skjulte evolutionære forbindelser. Men indtil fremkomsten af værktøjer som AlphaFold, der kan forudsige proteinstrukturer i stor skala, var det ikke muligt at sammenligne proteinstrukturer på tværs af en hel familie af vira, siger Joe Grove, en molekylær virolog ved University of Glasgow, UK.
I en artikel offentliggjort i denne uge iNatur 1Grove og hans team demonstrerer styrken af en strukturbaseret tilgang til flavivirus - en gruppe, der inkluderer hepatitis C, dengue- og Zika-vira samt adskillige vigtige dyrepatogener og -arter, der kan udgøre nye trusler mod menneskers sundhed.
Hvordan vira invaderer
Forskernes forståelse af flavivirus-evolution er primært baseret på sekvenser af langsomt udviklende enzymer, der kopierer deres genetiske materiale. Der er dog bemærkelsesværdigt lidt kendt om oprindelsen af de "virale indgangsproteiner", som flavivirus bruger til at trænge ind i celler, og som bestemmer værten, de kan inficere. Grove hævder, at denne videnskløft vil hindre udviklingen af en effektiv vaccine mod Hepatitis C, som dræber hundredtusindvis af mennesker hvert år.
"På sekvensniveau er tingene så divergerende, at vi ikke kan sige, om de er beslægtede eller ej," siger han. "Gennembruddet i forudsigelse af proteinstruktur åbner hele spørgsmålet, og vi kan se tingene ret klart."
Forskerne brugte DeepMinds AlphaFold2 -model og ESMFold, en Struktur- Forudsigelsesværktøj udviklet af teknologigiganten Meta, for at generere mere end 33.000 forudsagte strukturer for proteiner fra 458 flavivirusarter. ESMFold er baseret på en sprogmodel trænet med titusinder af proteinsekvenser. I modsætning til AlphaFold kræver det kun én inputsekvens i stedet for at stole på flere sekvenser af lignende proteiner, hvilket kunne gøre det særligt nyttigt til at studere de mest mystiske vira.
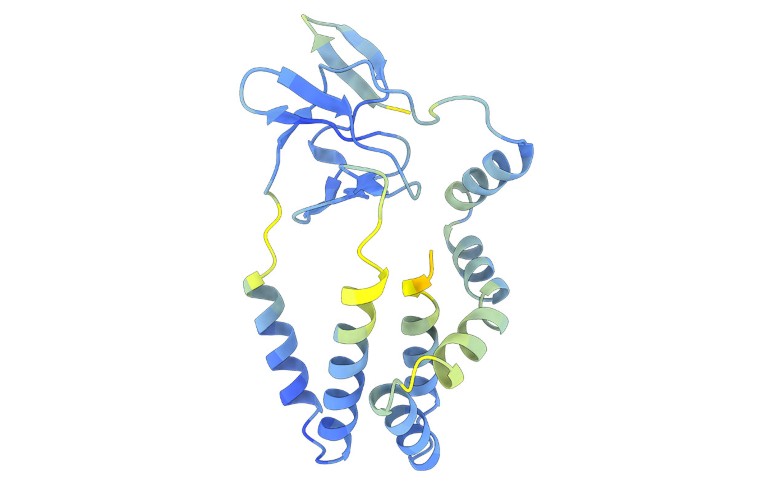
De forudsagte strukturer gjorde det muligt for forfatterne at identificere virale indgangsproteiner, hvis sekvenser adskiller sig meget fra dem af kendte flavivira. De fandt nogle uventede forbindelser. Så gruppen af vira, der inkluderer hepatitis C, bruger et system til at inficere celler, der ligner det, de opdagede med pestivira - en gruppe, der inkluderer den klassiske svineinfluenzavirus, som forårsager hæmoragisk feber hos grise og andre dyrepatogener.
De AI-drevne sammenligninger viste, at dette inputsystem er forskelligt fra mange andre flavivira. "For hepatitis C og dens pårørende ved vi ikke, hvor deres indgangssystem kommer fra. Det kunne være opfundet," siger Grove.
Stjålet af bakterier
De forudsagte strukturer viste også, at de velundersøgte inputproteiner fra Zika- og dengue-vira har samme oprindelse som dem fra de "underlige og vidunderlige" flavivira med enorme genomer, herunder Haseki-flåtvirus, som kan give feber hos mennesker. En anden stor overraskelse var opdagelsen af, at nogle flavivirus besidder et enzym, der ser ud til at være blevet stjålet fra bakterier.
"Dette ville være uden fortilfælde," siger virolog Mary Petrone fra University of Sydney, Australien, hvis det ikke var for hendes holds opdagelse i år af et lignende tyveri af en særlig "underlig og vidunderlig" art af flavivirus 2. "Genetisk piratkopiering kan have spillet en større rolle i udviklingen af flavivirus end tidligere antaget," tilføjer hun.
David Moi, en beregningsbiolog ved University of Lausanne, Schweiz, siger, at flavivirusundersøgelsen kun er toppen af isbjerget, og at de evolutionære historier om andre vira og endda nogle cellulære organismer sandsynligvis vil blive genfortalt ved hjælp af AI. "Nu hvor vi kan kigge videre, skal alle disse ting have en lille opdatering," siger han.

 Suche
Suche
 Mein Konto
Mein Konto