L’intelligence artificielle (IA) contribue à redessiner l’arbre généalogique des virus. Structures protéiques prédites à l’aide AlphaFold et ceux inspirés des chatbots « Modèles de langage protéique » ont découvert des liens surprenants dans une famille de virus qui comprend des agents pathogènes qui infectent les humains et des menaces émergentes.
Une grande partie de la compréhension des scientifiques sur évolution virale est basé sur la comparaison des génomes. Cependant, l’évolution ultra-rapide des virus, notamment ceux dotés d’un génome à ARN, et leur propension à acquérir du matériel génétique provenant d’autres organismes montre que les séquences génétiques peuvent cacher des relations plus profondes et plus distantes entre virus, qui peuvent varier selon le gène étudié.
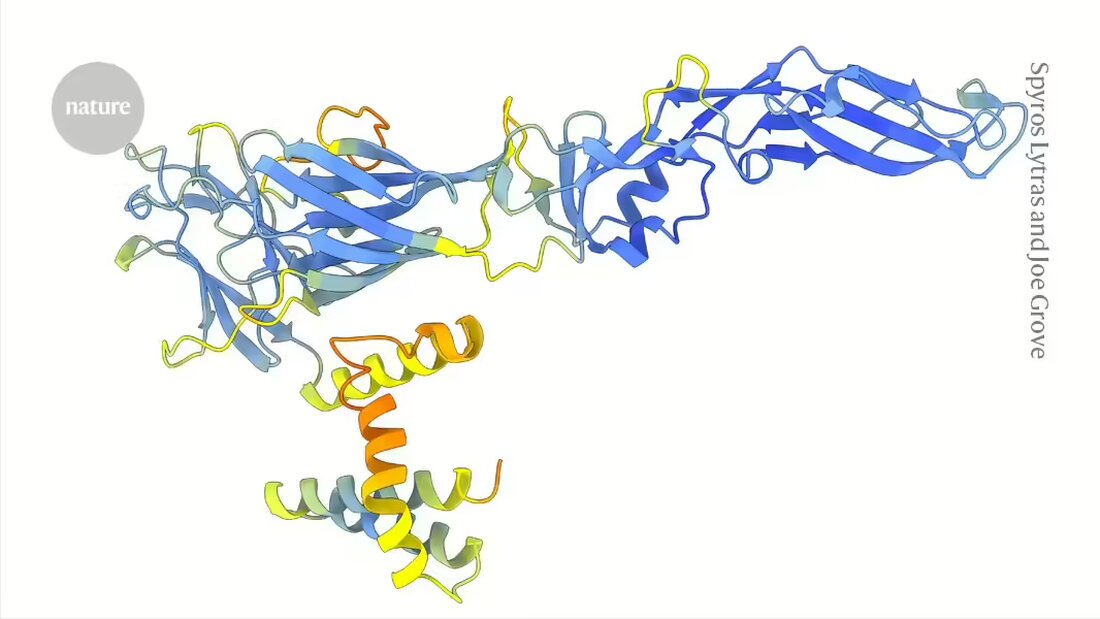
En revanche, les formes ou structures des protéines codées par les gènes viraux ont tendance à changer lentement, ce qui permet de détecter ces connexions évolutives cachées. Cependant, jusqu'à l'avènement d'outils comme AlphaFold, capables de prédire les structures protéiques à grande échelle, il n'était pas possible de comparer les structures protéiques d'une famille entière de virus, explique Joe Grove, virologue moléculaire à l'Université de Glasgow, au Royaume-Uni.
Dans un article publié cette semaine dansNature 1Grove et son équipe démontrent la puissance d'une approche basée sur la structure des flavivirus, un groupe qui comprend les virus de l'hépatite C, de la dengue et du Zika, ainsi que plusieurs agents pathogènes animaux importants et espèces susceptibles de constituer des menaces émergentes pour la santé humaine.
Comment les virus envahissent
La compréhension des chercheurs sur l'évolution des flavivirus repose principalement sur des séquences d'enzymes à évolution lente qui copient leur matériel génétique. Cependant, on sait remarquablement peu de choses sur les origines des protéines « d’entrée virale » que les flavivirus utilisent pour pénétrer dans les cellules et qui déterminent l’hôte qu’ils peuvent infecter. Grove affirme que ce manque de connaissances entravera le développement d'un vaccin efficace contre Hépatite C, qui tue des centaines de milliers de personnes chaque année.
« Au niveau des séquences, les choses sont tellement divergentes qu’on ne peut pas dire si elles sont liées ou non », dit-il. "La percée dans la prédiction de la structure des protéines ouvre toute la question, et nous pouvons voir les choses assez clairement."
Les chercheurs ont utilisé Esprits profonds AlphaFold2 -modèle et ESMFold, un Structure- Outil de prédiction développé par le géant de la technologie Meta, pour générer plus de 33 000 structures prédites pour les protéines de 458 espèces de flavivirus. ESMFold est basé sur un modèle de langage entraîné avec des dizaines de millions de séquences protéiques. Contrairement à AlphaFold, il ne nécessite qu’une seule séquence d’entrée plutôt que de s’appuyer sur plusieurs séquences de protéines similaires, ce qui pourrait le rendre particulièrement utile pour étudier les virus les plus mystérieux.
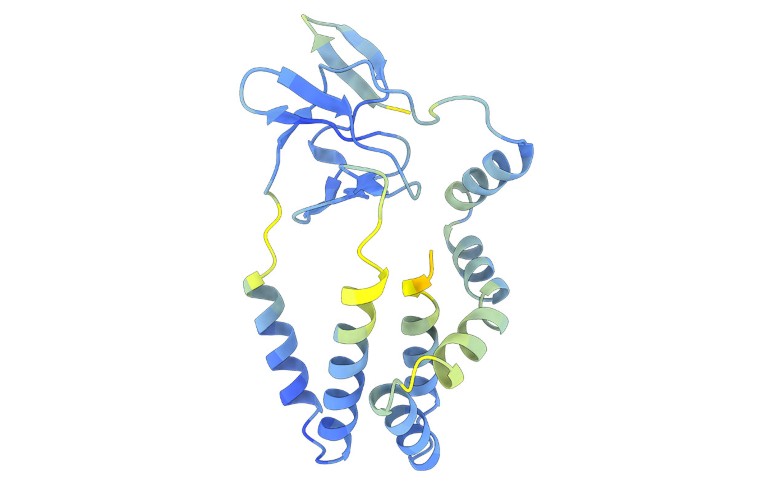
Les structures prédites ont permis aux auteurs d’identifier des protéines d’entrée virales dont les séquences diffèrent grandement de celles des flavivirus connus. Ils ont trouvé des liens inattendus. Ainsi, le groupe de virus qui comprend l’hépatite C utilise un système pour infecter les cellules similaire à celui qu’ils ont découvert avec les pestivirus – un groupe qui comprend le virus classique de la grippe porcine, qui provoque une fièvre hémorragique chez les porcs, et d’autres agents pathogènes animaux.
Les comparaisons basées sur l’IA ont montré que ce système d’entrée est différent de celui de nombreux autres flavivirus. "Pour l'hépatite C et ses proches, nous ne savons pas d'où vient leur système d'entrée. Il aurait pu être inventé", explique Grove.
Volé par des bactéries
Les structures prédites ont également montré que les protéines d'entrée bien étudiées des virus Zika et de la dengue ont les mêmes origines que celles des flavivirus "étranges et merveilleux" dotés d'énormes génomes, y compris le virus de la tique Haseki, qui peut provoquer de la fièvre chez l'homme. Une autre grande surprise a été la découverte que certains flavivirus possèdent une enzyme qui semble avoir été volée à des bactéries.
"Cela serait sans précédent", déclare la virologue Mary Petrone de l'Université de Sydney, en Australie, si son équipe n'avait découvert cette année un vol similaire d'une espèce particulièrement "étrange et merveilleuse" de flavivirus. 2. "Le piratage génétique a peut-être joué un rôle plus important dans l'évolution des flavivirus qu'on ne le pensait auparavant", ajoute-t-elle.
David Moi, biologiste informatique à l'Université de Lausanne, en Suisse, affirme que l'étude sur les flavivirus n'est que la pointe de l'iceberg et que les histoires évolutives d'autres virus et même de certains organismes cellulaires seront probablement racontées à l'aide de l'IA. "Maintenant que nous pouvons aller plus loin, toutes ces choses doivent faire l'objet d'une petite mise à jour", dit-il.

 Suche
Suche
 Mein Konto
Mein Konto