Tekoäly (AI) auttaa piirtämään uudelleen virusten sukupuuta. Ennustetut proteiinirakenteet käyttämällä AlphaFold ja chatbot-vaikutteisia "Proteiinikielimallit" ovat löytäneet yllättäviä yhteyksiä virusperheessä, joka sisältää taudinaiheuttajia, jotka tartuttavat ihmisiä ja uusia uhkia.
Suuri osa tutkijoiden ymmärryksestä viruksen evoluutio perustuu genomien vertailuun. Kuitenkin virusten, erityisesti niiden, joilla on RNA-genomeja, salamannopea evoluutio ja niiden taipumus hankkia geneettistä materiaalia muista organismeista osoittaa, että geneettiset sekvenssit voivat piilottaa syvempiä ja etäisempiä virusten välisiä suhteita, jotka voivat vaihdella tutkittavan geenin mukaan.
Sitä vastoin virusgeenien koodaamien proteiinien muodot tai rakenteet pyrkivät muuttumaan hitaasti, mikä tekee mahdolliseksi havaita nämä piilotetut evoluutioyhteydet. Kuitenkin ennen AlphaFoldin kaltaisten työkalujen tuloa, jotka voivat ennustaa proteiinien rakenteita suuressa mittakaavassa, ei ollut mahdollista verrata proteiinirakenteita koko virusperheessä, sanoo Joe Grove, molekyylivirologi Glasgow'n yliopistosta, Yhdistyneestä kuningaskunnasta.
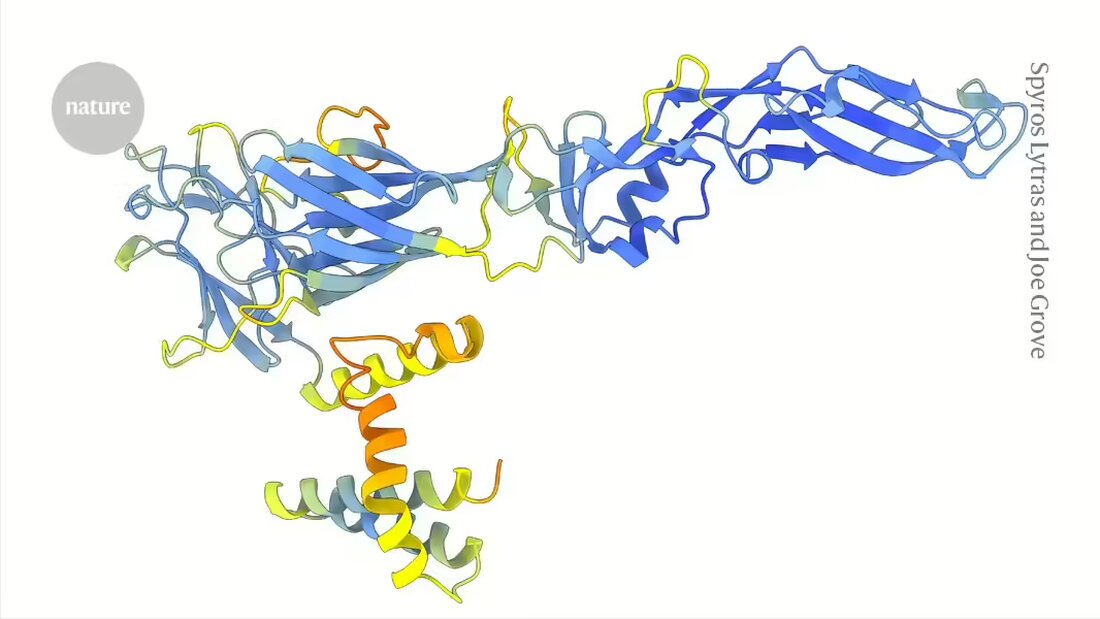
Tällä viikolla julkaistussa artikkelissaLuonto 1Grove ja hänen tiiminsä osoittavat rakenteeseen perustuvan lähestymistavan tehon flaviviruksiin – ryhmään, johon kuuluvat hepatiitti C-, dengue- ja Zika-virukset sekä useita tärkeitä eläinpatogeenejä ja lajeja, jotka voivat aiheuttaa uusia uhkia ihmisten terveydelle.
Miten virukset hyökkäävät
Tutkijoiden käsitys flaviviruksen evoluutiosta perustuu ensisijaisesti hitaasti kehittyvien entsyymien sekvensseihin, jotka kopioivat niiden geneettistä materiaalia. Kuitenkin huomattavan vähän tiedetään niiden "viruksen sisäänpääsyn" proteiinien alkuperästä, joita flavivirukset käyttävät päästäkseen soluihin ja jotka määrittävät isännän, jonka ne voivat infektoida. Grove väittää, että tämä tiedon puute estää tehokkaan rokotteen kehittämistä vastaan C-hepatiitti, joka tappaa satoja tuhansia ihmisiä joka vuosi.
"Sekvenssitasolla asiat ovat niin erilaisia, että emme voi sanoa, liittyvätkö ne toisiinsa vai eivät", hän sanoo. "Proteiinirakenteen ennustamisen läpimurto avaa koko kysymyksen, ja voimme nähdä asiat melko selvästi."
Tutkijat käyttivät DeepMinds AlphaFold2 - malli ja ESMFold, a Rakenne- Teknologiajätti Metan kehittämä ennustustyökalu, tuottaa yli 33 000 ennustettua rakennetta proteiineille 458 flaviviruslajista. ESMFold perustuu kielimalliin, joka on koulutettu kymmenillä miljoonilla proteiinisekvensseillä. Toisin kuin AlphaFold, se vaatii vain yhden syöttösekvenssin sen sijaan, että se luottaisi useisiin samankaltaisten proteiinien sekvensseihin, mikä voisi tehdä siitä erityisen hyödyllisen salaperäisimpien virusten tutkimiseen.
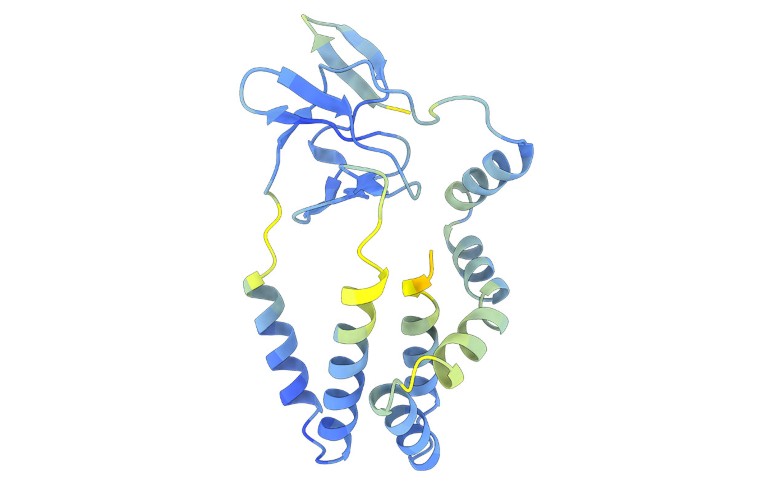
Ennustetut rakenteet antoivat kirjoittajille mahdollisuuden tunnistaa viruksen sisäänpääsyproteiineja, joiden sekvenssit eroavat suuresti tunnettujen flavivirusten sekvenssistä. He löysivät odottamattomia yhteyksiä. Joten virusryhmä, johon kuuluu C-hepatiitti, käyttää järjestelmää infektoimaan soluja, jotka ovat samankaltaisia kuin pestiviruksilla – ryhmä, johon kuuluu klassinen sikainfluenssavirus, joka aiheuttaa verenvuotokuumetta sioilla, ja muita eläinpatogeenejä.
Tekoälykäyttöiset vertailut osoittivat, että tämä syöttöjärjestelmä eroaa monista muista flaviviruksista. "Hepatiitti C:n ja sen sukulaisten kohdalla emme tiedä, mistä heidän pääsyjärjestelmänsä tulee. Se olisi voitu keksiä", Grove sanoo.
Bakteerien varastama
Ennustetut rakenteet osoittivat myös, että Zika- ja dengue-virusten hyvin tutkituilla syöteproteiineilla on sama alkuperä kuin "oudoilla ja ihmeellisillä" flaviviruksilla, joilla on valtava genomi, mukaan lukien Haseki-punkkivirus, joka voi aiheuttaa kuumetta ihmisillä. Toinen suuri yllätys oli löytö, että joillakin flaviviruksilla on entsyymi, joka näyttää varastetun bakteereista.
"Tämä olisi ennennäkemätöntä", sanoo virologi Mary Petrone Sydneyn yliopistosta Australiasta, ellei hänen tiiminsä olisi tänä vuonna löytänyt samanlaista erityisen "outoa ja ihmeellistä" flaviviruslajia. 2. "Geneettisellä piratismilla on saattanut olla suurempi rooli flavivirusten evoluutiossa kuin aiemmin uskottiin", hän lisää.
Laskennallinen biologi David Moi Lausannen yliopistosta Sveitsistä sanoo, että flavivirustutkimus on vain jäävuoren huippu ja että muiden virusten ja jopa joidenkin solueliöiden evoluutiotarinat todennäköisesti kerrotaan uudelleen tekoälyn avulla. "Nyt kun voimme katsoa pidemmälle, kaikkia näitä asioita on päivitettävä hieman", hän sanoo.

 Suche
Suche
 Mein Konto
Mein Konto