La inteligencia artificial (IA) está ayudando a rediseñar el árbol genealógico de los virus. Estructuras proteicas predichas usando AlfaFold y otros inspirados en chatbots “Modelos de lenguaje proteico” han descubierto conexiones sorprendentes en una familia de virus que incluye patógenos que infectan a los humanos y amenazas emergentes.
Una gran parte de la comprensión de los científicos sobre evolución viral Se basa en la comparación de genomas. Sin embargo, la rápida evolución de los virus, particularmente aquellos con genomas de ARN, y su propensión a adquirir material genético de otros organismos muestra que las secuencias genéticas pueden ocultar relaciones más profundas y distantes entre los virus, que pueden variar según el gen que se estudie.
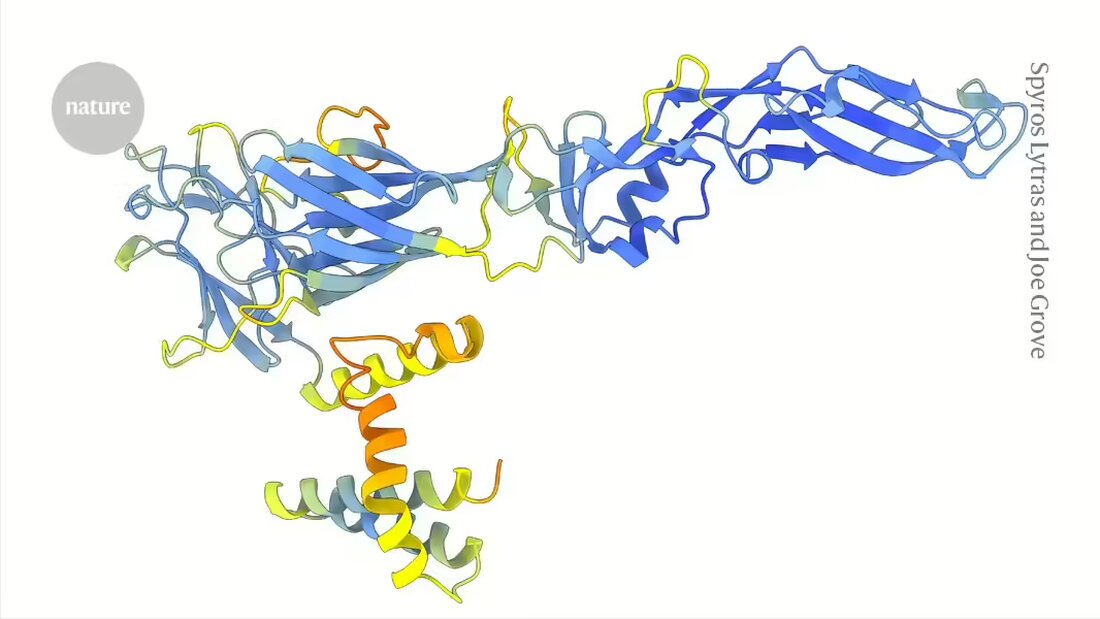
Por el contrario, las formas o estructuras de las proteínas codificadas por genes virales tienden a cambiar lentamente, lo que permite detectar estas conexiones evolutivas ocultas. Sin embargo, hasta la llegada de herramientas como AlphaFold, que pueden predecir estructuras de proteínas a gran escala, no era posible comparar estructuras de proteínas en toda una familia de virus, afirma Joe Grove, virólogo molecular de la Universidad de Glasgow, Reino Unido.
En un artículo publicado esta semana enNaturaleza 1Grove y su equipo demuestran el poder de un enfoque basado en la estructura de los flavivirus, un grupo que incluye los virus de la hepatitis C, el dengue y el Zika, así como varios patógenos animales importantes y especies que pueden representar amenazas emergentes para la salud humana.
Cómo invaden los virus
La comprensión de los investigadores sobre la evolución de los flavivirus se basa principalmente en secuencias de enzimas de evolución lenta que copian su material genético. Sin embargo, se sabe muy poco sobre los orígenes de las proteínas de “entrada viral” que utilizan los flavivirus para ingresar a las células y que determinan el huésped que pueden infectar. Grove sostiene que esta brecha de conocimiento obstaculizará el desarrollo de una vacuna eficaz contra Hepatitis C, que mata a cientos de miles de personas cada año.
"A nivel de secuencia, las cosas son tan divergentes que no podemos decir si están relacionadas o no", dice. "El gran avance en la predicción de la estructura de las proteínas abre toda la cuestión y podemos ver las cosas con bastante claridad".
Los investigadores utilizaron Mentes profundas AlfaFold2 -modelo y ESMFold, un Estructura- Herramienta de predicción desarrollada por el gigante tecnológico Meta, para generar más de 33.000 estructuras previstas para proteínas de 458 especies de flavivirus. ESMFold se basa en un modelo de lenguaje entrenado con decenas de millones de secuencias de proteínas. A diferencia de AlphaFold, solo requiere una secuencia de entrada en lugar de depender de múltiples secuencias de proteínas similares, lo que podría hacerlo particularmente útil para estudiar los virus más misteriosos.
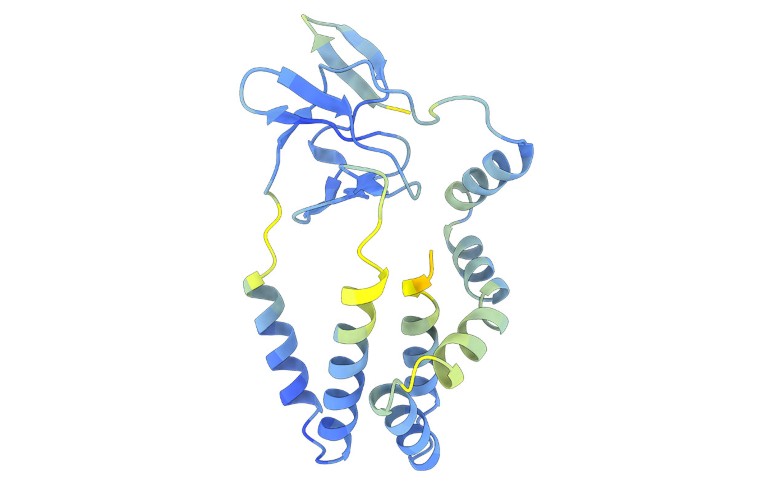
Las estructuras predichas permitieron a los autores identificar proteínas de entrada viral cuyas secuencias difieren mucho de las de los flavivirus conocidos. Encontraron algunas conexiones inesperadas. Entonces, el grupo de virus que incluye la hepatitis C utiliza un sistema para infectar células similar al que descubrieron con los pestivirus, un grupo que incluye el virus clásico de la gripe porcina, que causa fiebre hemorrágica en los cerdos, y otros patógenos animales.
Las comparaciones basadas en IA mostraron que este sistema de entrada es diferente al de muchos otros flavivirus. "En el caso de la hepatitis C y sus parientes, no sabemos de dónde viene su sistema de entrada. Podría haber sido inventado", afirma Grove.
Robado por bacterias
Las estructuras predichas también mostraron que las proteínas de entrada bien estudiadas de los virus Zika y del dengue tienen los mismos orígenes que las de los flavivirus "extraños y maravillosos" con genomas enormes, incluido el virus de la garrapata Haseki, que puede causar fiebre en humanos. Otra gran sorpresa fue el descubrimiento de que algunos flavivirus poseen una enzima que parece haber sido robada de las bacterias.
"Esto no tendría precedentes", afirma la viróloga Mary Petrone de la Universidad de Sydney, Australia, si no fuera por el descubrimiento este año por parte de su equipo de un robo similar de una especie de flavivirus particularmente "extraña y maravillosa". 2. "La piratería genética puede haber desempeñado un papel más importante de lo que se pensaba en la evolución de los flavivirus", añade.
David Moi, biólogo computacional de la Universidad de Lausana, Suiza, dice que el estudio de los flavivirus es sólo la punta del iceberg y que es probable que las historias evolutivas de otros virus e incluso de algunos organismos celulares se vuelvan a contar utilizando la IA. "Ahora que podemos mirar más allá, todas estas cosas necesitan una pequeña actualización", dice.

 Suche
Suche
 Mein Konto
Mein Konto