AI-proteinprediksjonsverktøy AlphaFold3 nå tilgjengelig som åpen kildekode
DeepMinds AlphaFold3 er nå åpen kildekode. Forskere kan bruke verktøyet for prediksjon av AI-proteinstruktur til ikke-kommersielle formål.
AI-proteinprediksjonsverktøy AlphaFold3 nå tilgjengelig som åpen kildekode
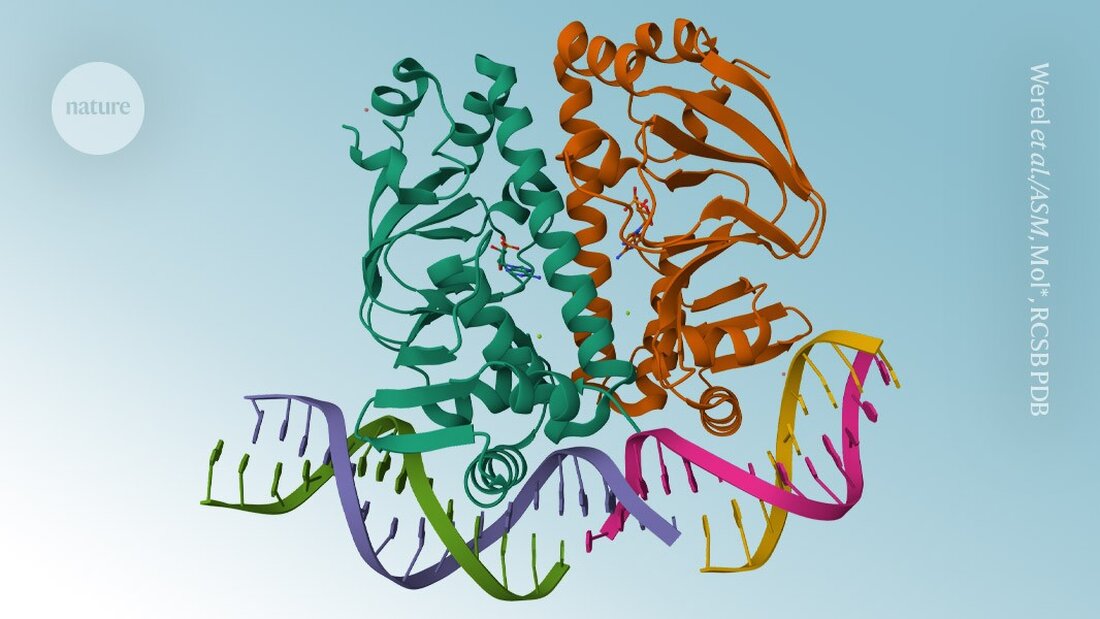
AlphaFold3 er endelig tilgjengelig. Seks måneder etter Google DeepMind kontroversielle koden en Artikler om prediksjonsmodellen for proteinstruktur forskere kan nå last ned programvarekoden og bruke verktøyet for kunstig intelligens for ikke-kommersielle applikasjoner, annonserte det London-baserte selskapet 11. november.
"Vi er veldig spente på å se hva folk gjør med det," sier John Jumper, som leder AlphaFold-teamet i DeepMind og begynte med administrerende direktør Demis Hassabis forrige måned vant en del av Nobelprisen i kjemi i 2024 for deres arbeid med AI-verktøyet.
I motsetning til forgjengerne, AlphaFold3 i stand til å modellere proteiner i kombinasjon med andre molekyler. I stedet for å frigi den underliggende koden - som tilfellet er med AlphaFold2 var tilfelle – DeepMind ga tilgang via en nettserver som begrenset antallet og typen spådommer forskerne kunne gjøre.
Kritisk nok tillot AlphaFold3-serveren ikke forskere å forutsi hvordan proteiner ville reagere i nærvær av potensielle medisiner. Men nå betyr DeepMinds beslutning om å gi ut koden at akademiske forskere kan forutsi slike interaksjoner ved å kjøre modellen selv.
Selskapet uttalte i utgangspunktet at å gjøre AlphaFold3 tilgjengelig kun via en webserver, var den rette balansen mellom å gi tilgang til forskning og å beskytte kommersielle ambisjoner. Isomorphic Labs, en spin-off av DeepMind i London, bruker AlphaFold3 i legemiddeloppdagelse.
Imidlertid trakk utgivelsen av AlphaFold3 uten kode eller modellvekter — Parametre oppnådd ved å trene programvaren på proteinstrukturer og andre data — fikk kritikk fra forskere som sa at flyttingen undergravde reproduserbarheten. DeepMind trakk raskt konklusjoner og sa at en åpen kildekode-versjon av verktøyet ville bli gjort tilgjengelig innen seks måneder.
Alle kan nå laste ned AlphaFold3-programvarekoden og bruke den ikke-kommersielt. Imidlertid har foreløpig kun forskere med akademisk tilknytning tilgang til treningsvektene på forespørsel.
Tilgjengelige versjoner
DeepMind har konkurranse: De siste månedene har flere selskaper Åpen kildekode-verktøy for prediksjon av proteinstruktur basert på AlphaFold3 presentert, som er avhengig av spesifikasjoner beskrevet i den originale artikkelen, kjent som pseudokode.
To kinesiske selskaper – teknologigiganten Baidu og TikTok-utvikleren ByteDance – har gitt ut sine egne AlphaFold3-inspirerte modeller, det samme har en oppstart i San Francisco, California kalt Chai Discovery.
En viktig ulempe med disse modellene er at ingen av dem, som AlphaFold3, er lisensiert for kommersielle applikasjoner som for eksempel medikamentoppdagelse, sier Mohammed AlQuraishi, en beregningsbiolog ved Columbia University i New York City. Chai Discoverys modell, Chai-1, kan imidlertid brukes til slikt arbeid via en webserver, forklarer Jack Dent, selskapets medgründer.
Et annet selskap, Ligo Biosciences i San Francisco, har gitt ut en restriksjonsfri versjon av AlphaFold3. Dette har imidlertid ennå ikke hele spekteret av funksjoner, inkludert muligheten til å modellere medikamenter og andre molekyler enn proteiner.
Andre team jobber med versjoner av AlphaFold3 som er tilgjengelige uten slike begrensninger: AlQuraishi håper å tilby en fullstendig åpen kildekode-modell kalt OpenFold3 senere i år. Dette vil tillate farmasøytiske selskaper å omskolere sine egne versjoner av modellen ved å bruke proprietære data, for eksempel strukturene til proteiner bundet til forskjellige legemidler, noe som potensielt kan forbedre ytelsen.
Åpenhet teller
Det siste året har det vært et rush av nye biologiske AI-modeller fra selskaper med ulike tilnærminger til åpenhet. Anthony Grid, en beregningsbiolog ved University of Wisconsin-Madison, har ingen problemer med at kommersielle selskaper kommer inn på feltet hans – så lenge de følger de samme reglene som andre forskere når de deler arbeidet sitt i tidsskrifter og preprint-servere.
Hvis DeepMind kommer med påstander om AlphaFold3 i en vitenskapelig publikasjon, "vil jeg forvente at de også deler informasjon om hvordan spådommene ble laget og gir AI-modellene og koden på en måte som vi kan teste dem," legger grid til. "Min gruppe vil ikke bruke verktøy som vi ikke kan verifisere."
Det faktum at flere replikasjoner av AlphaFold3 allerede har dukket opp viser at modellen var reproduserbar, selv uten åpen kildekode, sier Pushmeet Kohli, leder for AI for Science i DeepMind. Han legger til at han gjerne vil se mer diskusjon i fremtiden om publiseringsstandarder på et felt som i økende grad blir befolket av akademiske og bedriftsforskere.
Den åpne kildekoden til AlphaFold2 førte til en bølge av innovasjon fra andre forskere. For eksempel brukte vinnerne av en nylig proteinmodelleringskonkurranse AI-verktøyet til å designe nye proteiner som kan binde seg til et kreftmål. Jumpers favoritt AlphaFold2-hack kommer fra et team som brukte verktøyet til å identifisere et viktig protein som hjelper sædceller å feste seg til egg.
Jumper gleder seg til å se hvilke overraskelser som dukker opp etter utgivelsen av AlphaFold3 – selv om de ikke alltid lykkes. "Folk vil bruke det på merkelige måter," spår han. "Noen ganger vil det mislykkes, og noen ganger vil det lykkes."

 Suche
Suche
 Mein Konto
Mein Konto
