Lo strumento di previsione delle proteine AI AlphaFold3 è ora disponibile come open source
AlphaFold3 di DeepMind è ora open source. I ricercatori possono utilizzare lo strumento di previsione della struttura delle proteine AI per scopi non commerciali.
Lo strumento di previsione delle proteine AI AlphaFold3 è ora disponibile come open source
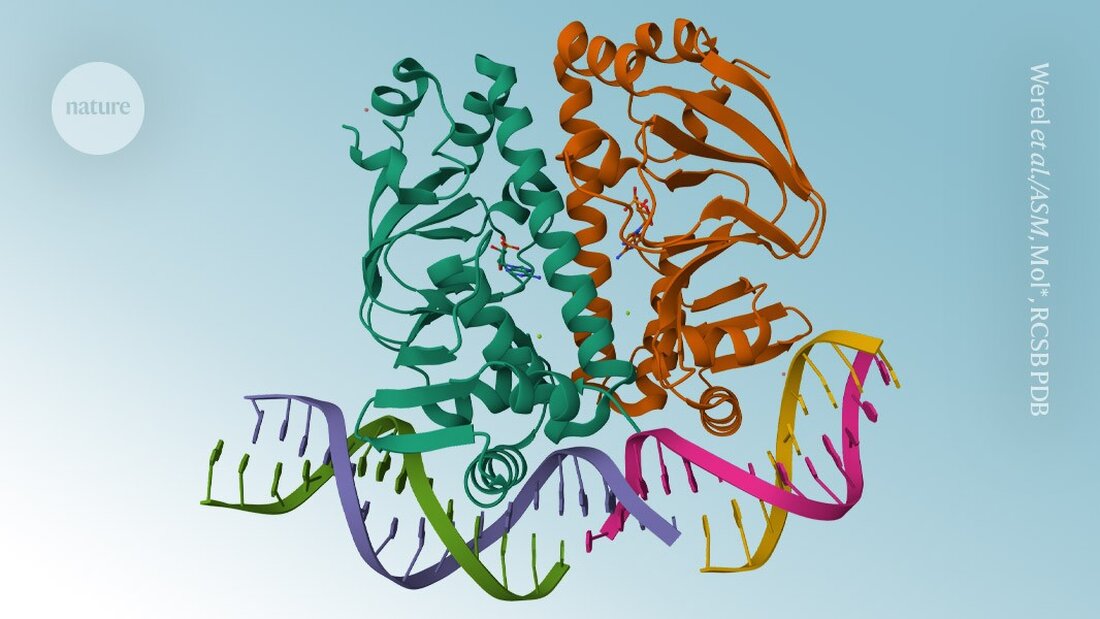
AlphaFold3 è finalmente disponibile. Sei mesi dopo Google DeepMind controverso il codice uno Articoli sul modello di previsione della struttura delle proteine gli scienziati ora possono scaricare il codice del software e utilizzare lo strumento di intelligenza artificiale per applicazioni non commerciali, ha annunciato l'11 novembre la società con sede a Londra.
"Siamo molto entusiasti di vedere cosa ne faranno le persone", afferma John Jumper, che guida il team AlphaFold presso DeepMind e si è unito al CEO Demis Hassabis il mese scorso ha vinto parte del Premio Nobel per la Chimica 2024 per il loro lavoro sullo strumento AI.
A differenza dei suoi predecessori, AlphaFold3 in grado di modellare proteine in combinazione con altre molecole. Invece di rilasciare il codice sottostante, come nel caso di AlphaFold2 è stato il caso: DeepMind ha fornito l’accesso tramite un server web che limitava il numero e il tipo di previsioni che gli scienziati potevano fare.
Fondamentalmente, il server AlphaFold3 non ha consentito agli scienziati di prevedere come le proteine avrebbero reagito in presenza di potenziali farmaci. Ma ora la decisione di DeepMind di rilasciare il codice significa che gli scienziati accademici possono prevedere tali interazioni eseguendo essi stessi il modello.
Inizialmente l'azienda aveva affermato che rendere disponibile AlphaFold3 solo tramite un server web rappresentava il giusto equilibrio tra la fornitura dell'accesso per la ricerca e la protezione delle ambizioni commerciali. Isomorphic Labs, uno spin-off di DeepMind a Londra, sta applicando AlphaFold3 alla scoperta di farmaci.
Tuttavia ha ritirato il rilascio di AlphaFold3 senza il codice o il peso del modello - I parametri ottenuti addestrando il software sulle strutture proteiche e altri dati - hanno attirato critiche da parte degli scienziati che hanno affermato che la mossa ha minato la riproducibilità. DeepMind ha tratto rapidamente le conclusioni e ha affermato che entro sei mesi sarà disponibile una versione open source dello strumento.
Chiunque può ora scaricare il codice del software AlphaFold3 e utilizzarlo a scopo non commerciale. Tuttavia, attualmente solo gli scienziati con affiliazioni accademiche hanno accesso ai pesi di formazione su richiesta.
Versioni accessibili
DeepMind ha concorrenza: negli ultimi mesi diverse aziende lo hanno fatto Presentati strumenti open source per la previsione della struttura delle proteine basati su AlphaFold3, che si basano sulle specifiche descritte nel documento originale, noto come pseudocodice.
Due aziende cinesi – il colosso tecnologico Baidu e lo sviluppatore TikTok ByteDance – hanno rilasciato i propri modelli ispirati ad AlphaFold3, così come una startup di San Francisco, in California, chiamata Chai Discovery.
Uno svantaggio principale di questi modelli è che nessuno di essi, come AlphaFold3, ha la licenza per applicazioni commerciali come la scoperta di farmaci, afferma Mohammed AlQuraishi, biologo computazionale della Columbia University di New York City. Tuttavia, il modello Chai-1 di Chai Discovery, può essere utilizzato per questo tipo di lavoro tramite un server web, spiega Jack Dent, cofondatore dell'azienda.
Un'altra società, Ligo Biosciences di San Francisco, ha rilasciato una versione senza restrizioni di AlphaFold3. Tuttavia, questo non ha ancora la gamma completa di funzioni, inclusa la capacità di modellare farmaci e molecole diverse dalle proteine.
Altri team stanno lavorando su versioni di AlphaFold3 disponibili senza tali restrizioni: AlQuraishi spera di offrire un modello completamente open source chiamato OpenFold3 entro la fine dell'anno. Ciò consentirebbe alle aziende farmaceutiche di riqualificare le proprie versioni del modello utilizzando dati proprietari, come le strutture delle proteine legate a diversi farmaci, migliorando potenzialmente le prestazioni.
L'apertura conta
L’anno scorso abbiamo assistito a un’ondata di nuovi modelli di intelligenza artificiale biologica da parte di aziende con diversi approcci all’apertura. Anthony Grid, biologo computazionale presso l’Università del Wisconsin-Madison, non ha problemi con l’ingresso di aziende commerciali nel suo campo, a patto che seguano le stesse regole degli altri scienziati quando condividono il loro lavoro su riviste e server di prestampa.
Se DeepMind facesse affermazioni su AlphaFold3 in una pubblicazione scientifica, "mi aspetterei che condividano anche informazioni su come sono state fatte le previsioni e forniscano modelli e codici di intelligenza artificiale in modo da poterli testare", aggiunge Grid. "Il mio gruppo non utilizzerà strumenti che non possiamo verificare."
Il fatto che siano già emerse diverse repliche di AlphaFold3 dimostra che il modello era riproducibile, anche senza codice open source, afferma Pushmeet Kohli, responsabile dell’AI for Science presso DeepMind. Aggiunge che gli piacerebbe vedere più discussioni in futuro sugli standard di pubblicazione in un campo sempre più popolato da ricercatori accademici e aziendali.
La natura open source di AlphaFold2 ha portato a un'ondata di innovazione da parte di altri scienziati. Ad esempio, i vincitori di un recente concorso di modellazione delle proteine hanno utilizzato lo strumento AI per per progettare nuove proteine che possano legarsi a un bersaglio del cancro. L'hack AlphaFold2 preferito di Jumper proviene da un team che ha utilizzato lo strumento per per identificare un’importante proteina che aiuta gli spermatozoi ad attaccarsi agli ovociti.
Jumper non vede l'ora di vedere quali sorprese emergeranno dopo il rilascio di AlphaFold3, anche se non sempre avranno successo. “Le persone lo useranno in modi strani”, prevede. “A volte fallirà e a volte avrà successo.”

 Suche
Suche
 Mein Konto
Mein Konto
