L'outil de prédiction des protéines d'IA AlphaFold3 est désormais disponible en open source
AlphaFold3 de DeepMind est désormais open source. Les chercheurs peuvent utiliser l’outil de prédiction de la structure des protéines de l’IA à des fins non commerciales.
L'outil de prédiction des protéines d'IA AlphaFold3 est désormais disponible en open source
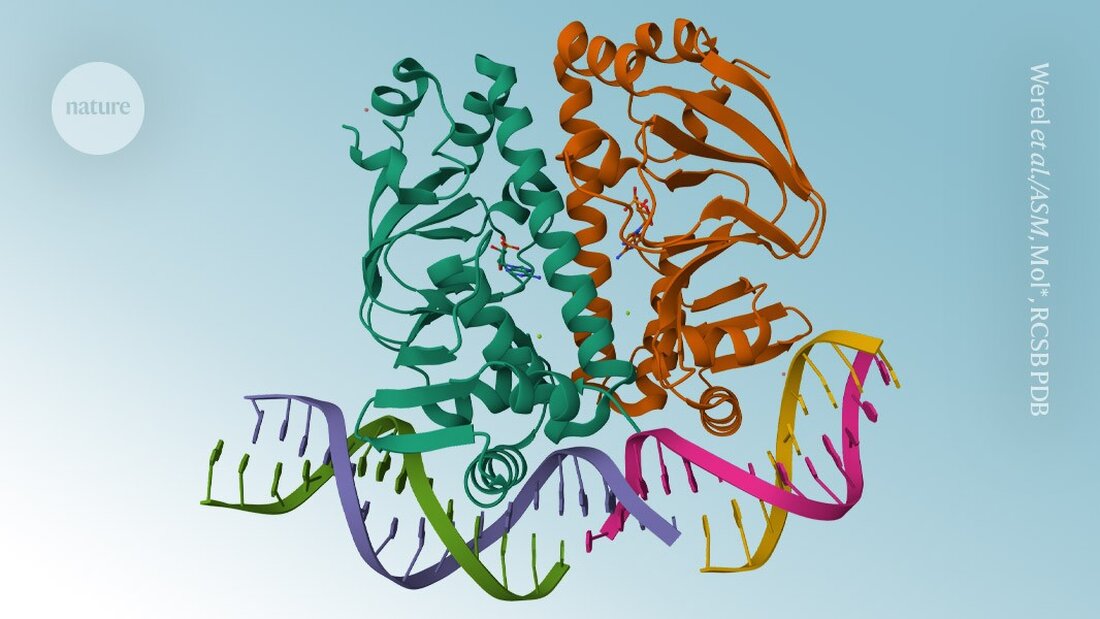
AlphaFold3 est enfin disponible. Six mois après Google DeepMind controversé le code un Articles sur le modèle de prédiction de la structure des protéines les scientifiques peuvent maintenant télécharger le code du logiciel et utiliser l'outil d'intelligence artificielle pour des applications non commerciales, a annoncé la société londonienne le 11 novembre.
"Nous sommes très impatients de voir ce que les gens en feront", déclare John Jumper, qui dirige l'équipe AlphaFold chez DeepMind et a rejoint le PDG Demis Hassabis le mois dernier. a remporté une partie du prix Nobel de chimie 2024 pour leur travail sur l'outil d'IA.
Contrairement à ses prédécesseurs, AlphaFold3 capable de modéliser des protéines en combinaison avec d’autres molécules. Au lieu de publier le code sous-jacent - comme c'est le cas avec AlphaFold2 C’était le cas – DeepMind fournissait un accès via un serveur Web qui limitait le nombre et le type de prédictions que les scientifiques pouvaient faire.
Il est important de noter que le serveur AlphaFold3 n’a pas permis aux scientifiques de prédire comment les protéines réagiraient en présence de médicaments potentiels. Mais maintenant, la décision de DeepMind de publier le code signifie que les scientifiques universitaires peuvent prédire de telles interactions en exécutant eux-mêmes le modèle.
La société a initialement déclaré que rendre AlphaFold3 disponible uniquement via un serveur Web représentait le bon équilibre entre fournir un accès à la recherche et protéger les ambitions commerciales. Isomorphic Labs, une spin-off de DeepMind à Londres, applique AlphaFold3 à la découverte de médicaments.
Cependant a retiré la sortie d'AlphaFold3 sans son code ni les poids du modèle - Les paramètres obtenus en entraînant le logiciel sur les structures protéiques et d'autres données - ont suscité les critiques des scientifiques qui ont déclaré que cette décision nuisait à la reproductibilité. DeepMind a rapidement tiré des conclusions et indiqué qu'une version open source de l'outil serait disponible d'ici six mois.
N’importe qui peut désormais télécharger le code du logiciel AlphaFold3 et l’utiliser à des fins non commerciales. Cependant, actuellement, seuls les scientifiques ayant des affiliations universitaires ont accès aux poids d’entraînement sur demande.
Versions accessibles
DeepMind a de la concurrence : ces derniers mois, plusieurs entreprises ont Présentation d'outils open source pour la prédiction de la structure des protéines basés sur AlphaFold3, qui s'appuient sur les spécifications décrites dans l'article original, connues sous le nom de pseudocode.
Deux sociétés chinoises – le géant de la technologie Baidu et le développeur de TikTok ByteDance – ont publié leurs propres modèles inspirés d'AlphaFold3, tout comme une startup de San Francisco, en Californie, appelée Chai Discovery.
Un inconvénient majeur de ces modèles est qu'aucun d'entre eux, comme AlphaFold3, n'est autorisé pour des applications commerciales telles que la découverte de médicaments, explique Mohammed AlQuraishi, biologiste computationnel à l'Université Columbia à New York. Cependant, le modèle Chai Discovery, Chai-1, peut être utilisé pour de tels travaux via un serveur Web, explique Jack Dent, co-fondateur de l'entreprise.
Une autre société, Ligo Biosciences de San Francisco, a publié une version sans restriction d'AlphaFold3. Cependant, celui-ci ne dispose pas encore de toutes les fonctions, notamment de la capacité de modéliser des médicaments et des molécules autres que les protéines.
D'autres équipes travaillent sur des versions d'AlphaFold3 disponibles sans de telles restrictions : AlQuraishi espère proposer un modèle entièrement open source appelé OpenFold3 plus tard cette année. Cela permettrait aux sociétés pharmaceutiques de recycler leurs propres versions du modèle en utilisant des données exclusives, telles que les structures des protéines liées à différents médicaments, améliorant ainsi potentiellement les performances.
L’ouverture compte
L’année dernière a été marquée par une ruée vers de nouveaux modèles d’IA biologique émanant d’entreprises ayant des approches différentes en matière d’ouverture. Anthony Grid, biologiste informatique à l'Université du Wisconsin-Madison, n'a aucun problème avec l'entrée d'entreprises commerciales dans son domaine, à condition qu'elles suivent les mêmes règles que les autres scientifiques lorsqu'elles partagent leurs travaux dans des revues et des serveurs de prépublication.
Si DeepMind fait des déclarations sur AlphaFold3 dans une publication scientifique, "je m'attendrais à ce qu'ils partagent également des informations sur la façon dont les prédictions ont été faites et fournissent les modèles et le code d'IA de manière à ce que nous puissions les tester", ajoute Grid. "Mon groupe n'utilisera pas d'outils que nous ne pouvons pas vérifier."
Le fait que plusieurs réplications d'AlphaFold3 aient déjà vu le jour montre que le modèle était reproductible, même sans code open source, explique Pushmeet Kohli, responsable de l'IA pour la science chez DeepMind. Il ajoute qu'il aimerait voir davantage de discussions à l'avenir sur les normes de publication dans un domaine de plus en plus peuplé de chercheurs universitaires et d'entreprises.
La nature open source d’AlphaFold2 a conduit à une vague d’innovation de la part d’autres scientifiques. Par exemple, les gagnants d'un récent concours de modélisation de protéines ont utilisé l'outil d'IA pour concevoir de nouvelles protéines capables de se lier à une cible cancéreuse. Le hack AlphaFold2 préféré de Jumper vient d'une équipe qui a utilisé l'outil pour identifier une protéine importante qui aide les spermatozoïdes à s'attacher aux ovules.
Jumper a hâte de voir quelles surprises surgiront après la sortie d'AlphaFold3 - même si elles ne sont pas toujours réussies. « Les gens l’utiliseront de façon étrange », prédit-il. « Parfois, cela échouera et parfois, cela réussira. »

 Suche
Suche
 Mein Konto
Mein Konto
