AI-proteinforudsigelsesværktøj AlphaFold3 nu tilgængelig som open source
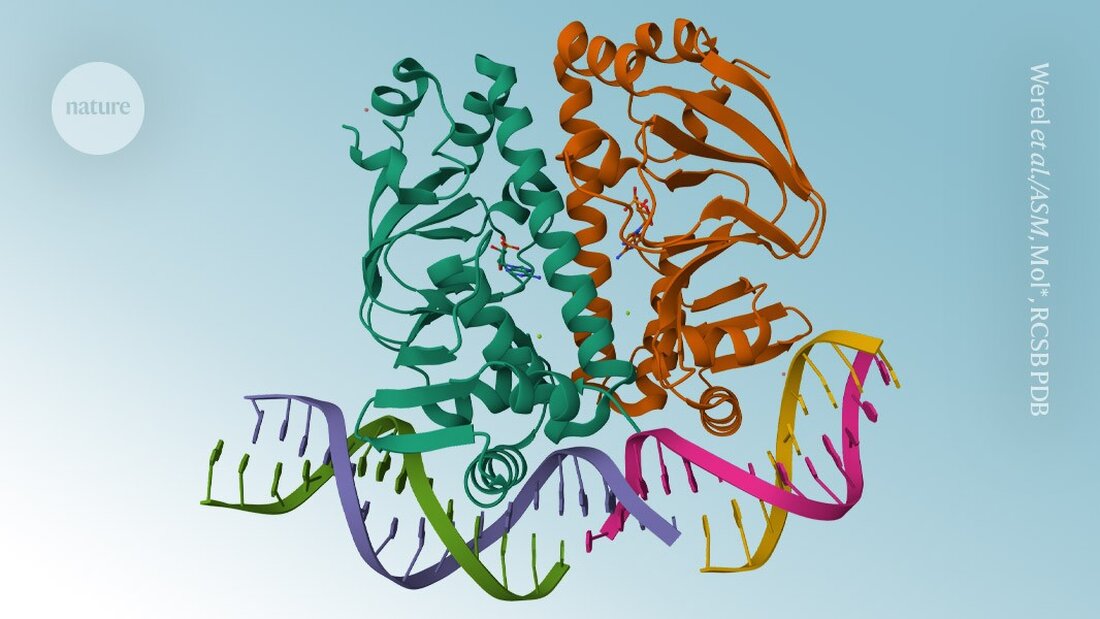
DeepMinds AlphaFold3 er nu open source. Forskere kan bruge værktøjet til forudsigelse af AI-proteinstruktur til ikke-kommercielle formål.
AI-proteinforudsigelsesværktøj AlphaFold3 nu tilgængelig som open source
AlphaFold3 er endelig tilgængelig. Seks måneder efter Google DeepMind kontroversielle koden en Paper om proteinstruktur-forudsigelsesmodellen det kan forskere nu download softwarekoden og bruge det kunstige intelligensværktøj til ikke-kommercielle applikationer, annoncerede det London-baserede firma den 11. november.
"Vi er meget spændte på at se, hvad folk gør med det," siger John Jumper, der leder AlphaFold-teamet hos DeepMind og sluttede sig til CEO Demis Hassabis i sidste måned vandt en del af Nobelprisen i kemi i 2024 for deres arbejde med AI-værktøjet.
I modsætning til sine forgængere, AlphaFold3 i stand til at modellere proteiner i kombination med andre molekyler. I stedet for at frigive den underliggende kode - som det er tilfældet med AlphaFold2 var tilfældet – DeepMind gav adgang via en webserver, der begrænsede antallet og typen af forudsigelser, videnskabsmænd kunne komme med.
Kritisk nok tillod AlphaFold3-serveren ikke videnskabsmænd at forudsige, hvordan proteiner ville reagere i nærvær af potentielle lægemidler. Men nu betyder DeepMinds beslutning om at frigive koden, at akademiske forskere kan forudsige sådanne interaktioner ved selv at køre modellen.
Virksomheden udtalte oprindeligt, at det at gøre AlphaFold3 kun tilgængeligt via en webserver, var den rette balance mellem at give adgang til forskning og beskytte kommercielle ambitioner. Isomorphic Labs, en spin-off af DeepMind i London, anvender AlphaFold3 til lægemiddelforskning.
Imidlertid trak udgivelsen af AlphaFold3 uden dens kode eller modelvægte — Parametre opnået ved at træne softwaren i proteinstrukturer og andre data — modtog kritik fra videnskabsmænd, der sagde, at flytningen underminerede reproducerbarheden. DeepMind trak hurtigt konklusioner og sagde, at en open source-version af værktøjet ville blive gjort tilgængelig inden for seks måneder.
Alle kan nu downloade AlphaFold3-softwarekoden og bruge den ikke-kommercielt. Men i øjeblikket er det kun forskere med akademisk tilhørsforhold, der har adgang til træningsvægtene efter anmodning.
Tilgængelige versioner
DeepMind har konkurrence: I de seneste måneder har flere virksomheder Open source værktøjer til forudsigelse af proteinstruktur baseret på AlphaFold3 præsenteret, som er afhængige af specifikationer beskrevet i det originale papir, kendt som pseudokode.
To kinesiske virksomheder - teknologigiganten Baidu og TikTok-udvikleren ByteDance - har udgivet deres egne AlphaFold3-inspirerede modeller, ligesom en startup i San Francisco, Californien, kaldet Chai Discovery.
En vigtig ulempe ved disse modeller er, at ingen af dem, ligesom AlphaFold3, er licenseret til kommercielle applikationer såsom opdagelse af lægemidler, siger Mohammed AlQuraishi, en beregningsbiolog ved Columbia University i New York City. Chai Discoverys model, Chai-1, kan dog bruges til sådan arbejde via en webserver, forklarer Jack Dent, virksomhedens medstifter.
Et andet firma, Ligo Biosciences fra San Francisco, har udgivet en begrænsningsfri version af AlphaFold3. Dette har dog endnu ikke hele rækken af funktioner, herunder evnen til at modellere andre lægemidler og molekyler end proteiner.
Andre teams arbejder på versioner af AlphaFold3, der er tilgængelige uden sådanne begrænsninger: AlQuraishi håber at tilbyde en fuldt ud open source-model kaldet OpenFold3 senere i år. Dette ville give medicinalvirksomheder mulighed for at genoptræne deres egne versioner af modellen ved hjælp af proprietære data, såsom strukturerne af proteiner bundet til forskellige lægemidler, hvilket potentielt kan forbedre ydeevnen.
Åbenhed tæller
Det seneste år har set en strøm af nye biologiske AI-modeller fra virksomheder med forskellige tilgange til åbenhed. Anthony Grid, en beregningsbiolog ved University of Wisconsin-Madison, har ingen problemer med, at kommercielle virksomheder kommer ind på hans felt - så længe de følger de samme regler som andre videnskabsmænd, når de deler deres arbejde i tidsskrifter og preprint-servere.
Hvis DeepMind fremsætter påstande om AlphaFold3 i en videnskabelig publikation, "vil jeg forvente, at de også deler information om, hvordan forudsigelserne blev lavet og leverer AI-modellerne og koden på en måde, så vi kan teste dem," tilføjer grid. "Min gruppe vil ikke bruge værktøjer, som vi ikke kan verificere."
Det faktum, at flere replikationer af AlphaFold3 allerede er dukket op, viser, at modellen var reproducerbar, selv uden åben kildekode, siger Pushmeet Kohli, leder af AI for Science hos DeepMind. Han tilføjer, at han gerne vil se mere diskussion i fremtiden om publiceringsstandarder på et felt, der i stigende grad befolkes af akademiske forskere og virksomheder.
AlphaFold2's open source-karakter førte til en bølge af innovation fra andre videnskabsmænd. For eksempel brugte vinderne af en nylig proteinmodelleringskonkurrence AI-værktøjet til at designe nye proteiner, der kan binde sig til et kræftmål. Jumpers foretrukne AlphaFold2-hack kommer fra et hold, der brugte værktøjet til at identificere et vigtigt protein, der hjælper sædceller med at binde sig til æg.
Jumper kan ikke vente med at se, hvilke overraskelser der dukker op efter udgivelsen af AlphaFold3 – også selvom de ikke altid er succesfulde. "Folk vil bruge det på mærkelige måder," forudser han. "Nogle gange vil det mislykkes, og nogle gange vil det lykkes."

 Suche
Suche
 Mein Konto
Mein Konto
