Nástroj pro predikci proteinů AI AlphaFold3 je nyní k dispozici jako open source
AlphaFold3 od DeepMind je nyní open source. Výzkumníci mohou použít nástroj pro predikci proteinové struktury AI pro nekomerční účely.
Nástroj pro predikci proteinů AI AlphaFold3 je nyní k dispozici jako open source
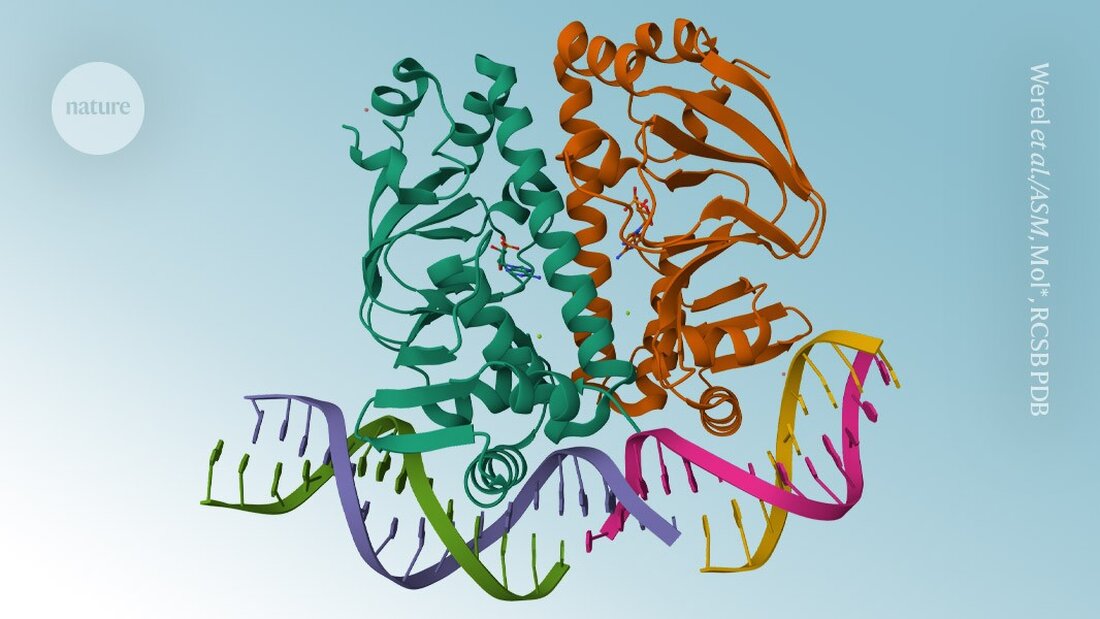
AlphaFold3 je konečně k dispozici. Šest měsíců po Google DeepMind kontroverzní kód jeden Referáty o modelu predikce struktury proteinů vědci nyní mohou stáhněte si softwarový kód a používat nástroj umělé inteligence pro nekomerční aplikace, oznámila londýnská společnost 11. listopadu.
„Velmi nás těší, že vidíme, co s tím lidé dělají,“ říká John Jumper, který vede tým AlphaFold v DeepMind a minulý měsíc se připojil k generálnímu řediteli Demis Hassabisovi. získal část Nobelovy ceny za chemii za rok 2024 za jejich práci na nástroji AI.
Na rozdíl od svých předchůdců, AlphaFold3 schopné modelovat proteiny v kombinaci s jinými molekulami. Místo uvolnění základního kódu - jak je tomu v případě AlphaFold2 byl tento případ – DeepMind poskytoval přístup přes webový server, který omezoval počet a typ předpovědí, které vědci mohli dělat.
Server AlphaFold3 kriticky neumožnil vědcům předpovědět, jak budou proteiny reagovat v přítomnosti potenciálních léků. Ale nyní rozhodnutí DeepMind uvolnit kód znamená, že akademičtí vědci mohou předvídat takové interakce tím, že sami spustí model.
Společnost původně uvedla, že zpřístupnění AlphaFold3 pouze prostřednictvím webového serveru vytvořilo správnou rovnováhu mezi poskytováním přístupu pro výzkum a ochranou komerčních ambicí. Isomorphic Labs, spin-off DeepMind v Londýně, používá AlphaFold3 při objevování léků.
Však vytáhl vydání AlphaFold3 bez jeho kódových nebo modelových vah — Parametry získané školením softwaru o proteinových strukturách a dalších datech — vyvolaly kritiku od vědců, kteří uvedli, že tento krok podkopal reprodukovatelnost. DeepMind rychle vyvodil závěry a uvedl, že open source verze nástroje bude zpřístupněna do šesti měsíců.
Kdokoli si nyní může stáhnout softwarový kód AlphaFold3 a používat jej nekomerčně. V současné době však mají přístup k tréninkovým závažím na vyžádání pouze vědci s akademickým vzděláním.
Přístupné verze
DeepMind má konkurenci: V posledních měsících má několik společností Prezentovány open source nástroje pro predikci struktury proteinů založené na AlphaFold3, které se opírají o specifikace popsané v původním článku, známé jako pseudokód.
Dvě čínské společnosti – technologický gigant Baidu a vývojář TikTok ByteDance – vydaly své vlastní modely inspirované AlphaFold3, stejně jako startup v San Franciscu v Kalifornii s názvem Chai Discovery.
Klíčovou nevýhodou těchto modelů je, že žádný z nich, jako AlphaFold3, není licencován pro komerční aplikace, jako je objevování léků, říká Mohammed AlQuraishi, počítačový biolog na Kolumbijské univerzitě v New Yorku. Nicméně model Chai Discovery, Chai-1, lze pro takovou práci použít prostřednictvím webového serveru, vysvětluje Jack Dent, spoluzakladatel společnosti.
Další společnost, Ligo Biosciences ze San Francisca, vydala verzi AlphaFold3 bez omezení. To však ještě nemá celou škálu funkcí, včetně schopnosti modelovat léky a molekuly jiné než proteiny.
Jiné týmy pracují na verzích AlphaFold3, které jsou dostupné bez takových omezení: AlQuraishi doufá, že koncem tohoto roku nabídne plně open-source model s názvem OpenFold3. To by farmaceutickým společnostem umožnilo přeškolit své vlastní verze modelu pomocí proprietárních dat, jako jsou struktury proteinů vázaných na různé léky, což by potenciálně zlepšilo výkon.
Otevřenost se počítá
Minulý rok byl svědkem návalu nových biologických modelů umělé inteligence od společností s různými přístupy k otevřenosti. Anthony Grid, počítačový biolog z University of Wisconsin-Madison, nemá problém se vstupem komerčních společností do jeho oboru – pokud dodržují stejná pravidla jako ostatní vědci při sdílení své práce v časopisech a na předtiskových serverech.
Pokud DeepMind tvrdí o AlphaFold3 ve vědecké publikaci, „očekával bych, že také budou sdílet informace o tom, jak byly předpovědi vytvořeny, a poskytnout modely AI a kód takovým způsobem, abychom je mohli otestovat,“ dodává grid. "Moje skupina nebude používat nástroje, které nemůžeme ověřit."
Skutečnost, že se již objevilo několik replikací AlphaFold3, ukazuje, že model byl reprodukovatelný i bez otevřeného zdrojového kódu, říká Pushmeet Kohli, vedoucí AI pro vědu v DeepMind. Dodává, že by si přál, aby se v budoucnu více diskutovalo o vydávání standardů v oblasti, která je stále více zastoupena akademickými a firemními výzkumníky.
Open source povaha AlphaFold2 vedla k prudkému nárůstu inovací od jiných vědců. Například vítězové nedávné soutěže v modelování proteinů použili nástroj AI navrhnout nové proteiny, které se mohou vázat na cíl rakoviny. Jumperův oblíbený hack AlphaFold2 pochází od týmu, který tento nástroj použil identifikovat důležitý protein, který pomáhá spermiím přichytit se k vajíčkům.
Jumper se nemůže dočkat, až uvidí, jaká překvapení se objeví po vydání AlphaFold3 - i když ne vždy jsou úspěšná. "Lidé to budou používat zvláštním způsobem," předpovídá. "Někdy to selže a někdy se to podaří."

 Suche
Suche
 Mein Konto
Mein Konto
